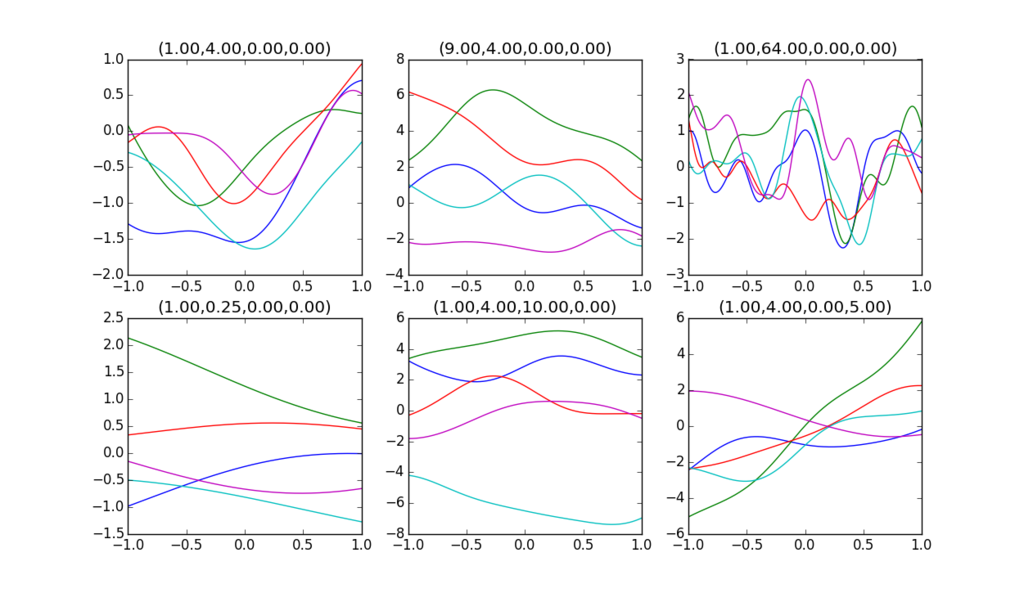
これです。
import numpy as np import matplotlib.pyplot as plt from numpy.random import multivariate_normal params = [(1,4,0,0), (9,4,0,0), (1,64,0,0), (1,0.25,0,0), (1,4,10,0), (1,4,0,5)] fig = plt.figure() for n in range(len(params)): (p0, p1, p2, p3) = params[n] linex = np.linspace(-1,1,999) kern = np.zeros([len(linex),len(linex)]) for i, x0 in enumerate(linex): for j, x1 in enumerate(linex): kern[i][j] = p0*np.exp(-p1*0.5*(x0-x1)*(x0-x1)) + p2 + p3*x0*x1 # kern[i][j] = np.exp(-p1*0.5*np.abs(x0-x1)) liney = multivariate_normal(np.zeros(len(linex)),kern,5) subplot = fig.add_subplot(2,3,n+1) subplot.set_title("(%1.2f,%1.2f,%1.2f,%1.2f)" % (p0,p1,p2,p3)) for c in range(5): subplot.plot(linex, liney[c]) plt.show()
説明
連続なグラフとして描いていますが、実際には多数の離散点 に対して乱数で
の値を決定しています。この際、
が N次元の多次元ガウス分布に従うという条件を設定しています。このような確率変数
をガウス過程と呼びます。
多次元ガウス分布なので、平均と分散共分散行列を指定すれば一意に定まりますが、ここでは特に、カーネル関数を次で定義した上で、
分散共分散行列を次式で決定しています。(平均は0に取ります。)
その上で、さまざまな に対してサンプルを取得したのが上記のグラフになります。
おまけ
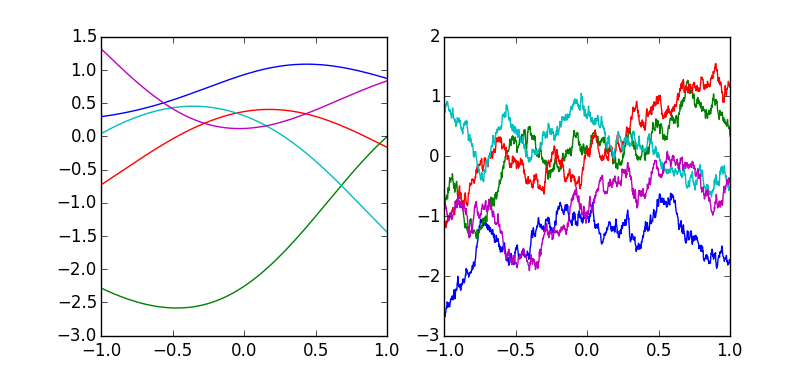
まったく同様に、Figure6.4は次のコードで再現できます。
import numpy as np import matplotlib.pyplot as plt from numpy.random import multivariate_normal fig = plt.figure() # Gaussian Kernel linex = np.linspace(-1,1,999) kern = np.zeros([len(linex),len(linex)]) for i, x0 in enumerate(linex): for j, x1 in enumerate(linex): kern[i][j] = np.exp(-0.5*(x0-x1)*(x0-x1)) liney = multivariate_normal(np.zeros(len(linex)),kern,5) subplot = fig.add_subplot(1,2,1) for c in range(5): subplot.plot(linex, liney[c]) # Exponential Kernel linex = np.linspace(-1,1,999) kern = np.zeros([len(linex),len(linex)]) for i, x0 in enumerate(linex): for j, x1 in enumerate(linex): kern[i][j] = np.exp(-0.5*np.abs(x0-x1)) liney = multivariate_normal(np.zeros(len(linex)),kern,5) subplot = fig.add_subplot(1,2,2) for c in range(5): subplot.plot(linex, liney[c]) plt.show()